require(tidyverse)
require(factoextra)
require(ggplot2)
require(ggdendro)Agrupamiento jerárquico en R
Recuerda que tenemos diversos posts sobre programación, pesquerías, ecología, entre otros. Te invitamos a revisar la lista en nuestra sección Posts.
Los métodos de agrupamiento (o clustering por su traducción al inglés) nos permiten agrupar observaciones similares. Existen diferentes formas de hacer esto, siendo una de las más utilizadas el agrupamiento jerárquico. En este tipo de agrupamiento, los grupos (o clusters) se pueden crean siguiendo una jerarquía de abajo hacia arriba (modo aglomerativo) o de arriba hacia abajo (modo divisivo). Una de las ventajas principales de este tipo de agrupamiento es que no tenemos que especificar un número de grupo a priori, como si lo requiere otros métodos como el k-means. Durante el proceso de construcción de los clusters se tienen que calcular medidas de distancia, los cuales nos ayudarán a determinar qué tan similares o diferentes son dos observaciones. Una de las medidas de distancia más utilizadas es la distancia Euclideana, aunque también se pueden usar: Manhattan, Bray Curtis, entre otras.
En este corto tutorial, explicaremos brevemente como realizar un agrupamiento jerárquico en R, cómo identificar el número óptimo de clusters, y cómo graficar nuestros resultados.
Comenzamos cargando las librerías que utilizaremos:
Primero pasamos a explorar las bases de datos que vamos a utilizar, la cual contiene información sobre la concentración de diferentes químicos presentes en vinos de una región de Italia, derivados de 3 diferentes cultivos. Los datos y más información sobre estos puede ser encontrados aquí.
Lo primero que haremos es leer la base de datos desde el sitio web, y luego lo transformamos a clase tibble:
my_data = read.table("http://archive.ics.uci.edu/ml/machine-learning-databases/wine/wine.data", sep=",")
my_data = my_data %>% as_tibble
head(my_data)# A tibble: 6 × 14
V1 V2 V3 V4 V5 V6 V7 V8 V9 V10 V11 V12 V13
<int> <dbl> <dbl> <dbl> <dbl> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 14.2 1.71 2.43 15.6 127 2.8 3.06 0.28 2.29 5.64 1.04 3.92
2 1 13.2 1.78 2.14 11.2 100 2.65 2.76 0.26 1.28 4.38 1.05 3.4
3 1 13.2 2.36 2.67 18.6 101 2.8 3.24 0.3 2.81 5.68 1.03 3.17
4 1 14.4 1.95 2.5 16.8 113 3.85 3.49 0.24 2.18 7.8 0.86 3.45
5 1 13.2 2.59 2.87 21 118 2.8 2.69 0.39 1.82 4.32 1.04 2.93
6 1 14.2 1.76 2.45 15.2 112 3.27 3.39 0.34 1.97 6.75 1.05 2.85
# ℹ 1 more variable: V14 <int>La primera columna nos indica el cultivo, y las otras 13 columnas es un químico diferente. Las filas son las muestras de vino utilizadas. Los valores mostrados son la concentración del químico respectivo para cada muestra.
dim(my_data)[1] 178 14Como vemos, en total tenemos 178 muestras. Ahora vamos a separar solo las 13 columnas con información de químicos, y vamos a calcular la distancia Euclideana entre cada muestra:
tmp_data = my_data[,2:14]
dist_data = dist(scale(tmp_data), method = "euclidean")dist_data contiene información de la distancia entre cada par de muestras. Obviamente, la distancia entre una misma muestra no es considerado. Ahora vamos a construir el agrupamiento jerárquico, para lo cual usaremos la función hclust:
my_clust = hclust(dist_data)El método de aglomeración por defecto es de tipo completo, pero existen otros como: ward.D2, single, average, etc. Te recomendamos explorarlos. Ahora vamos a graficar el agrupamiento utilizando la función ggdendrogram:
ggdendrogram(my_clust)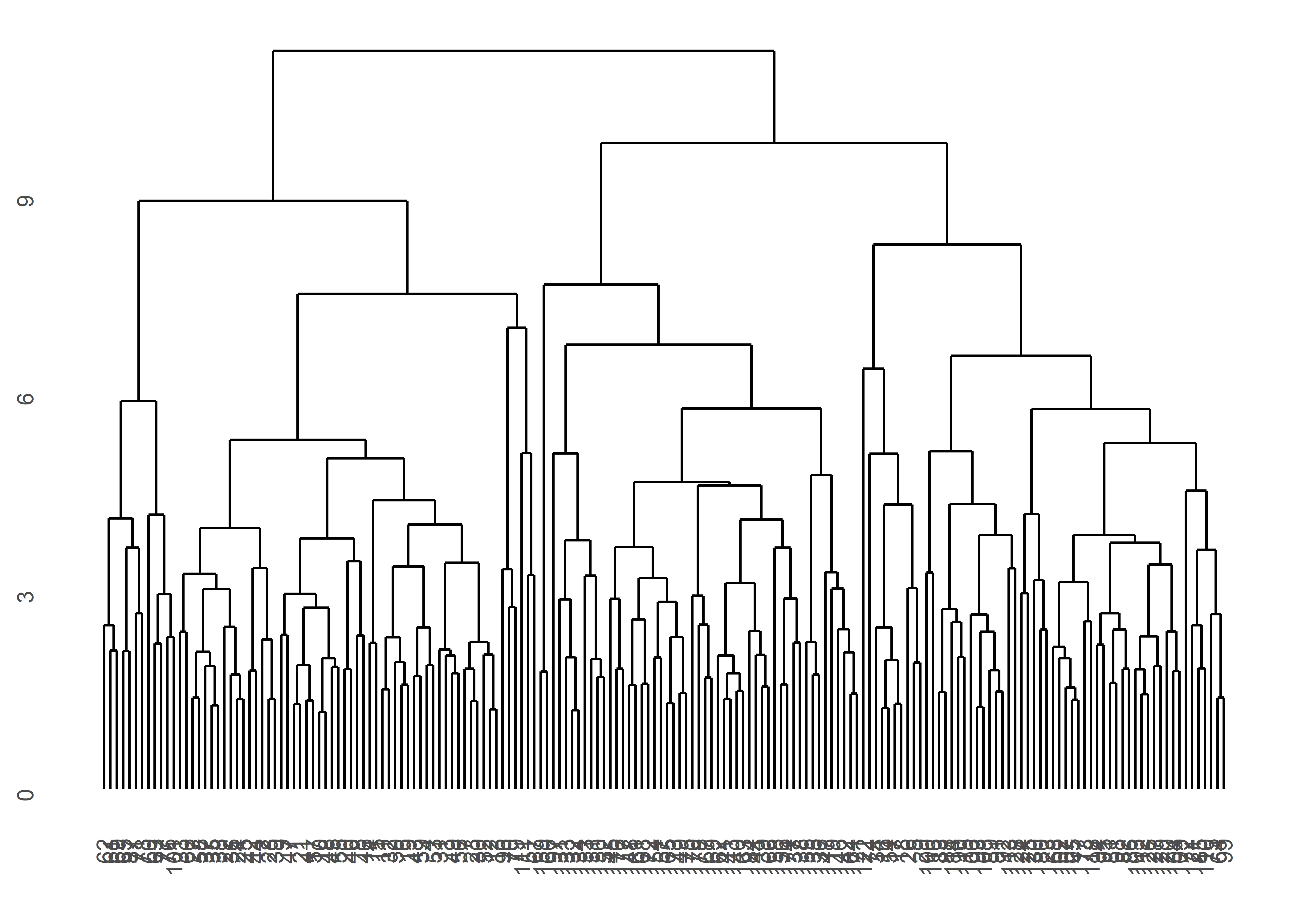
Como vemos, lo que hemos hecho es agrupar las muestras en diferentes clusters. Si vemos la gráfica de arriba hacia abajo, vemos que primero tenemos dos grupos claramente diferenciados, y luego cada uno de estos se divide en otros dos grupos.
Algo que resulta útil es identificar el número óptimo de clusters. Para hacer esto, utilizamos la función fviz_nbclust de la librería factoextra:
fviz_nbclust(tmp_data, FUN = hcut, method = 'silhouette')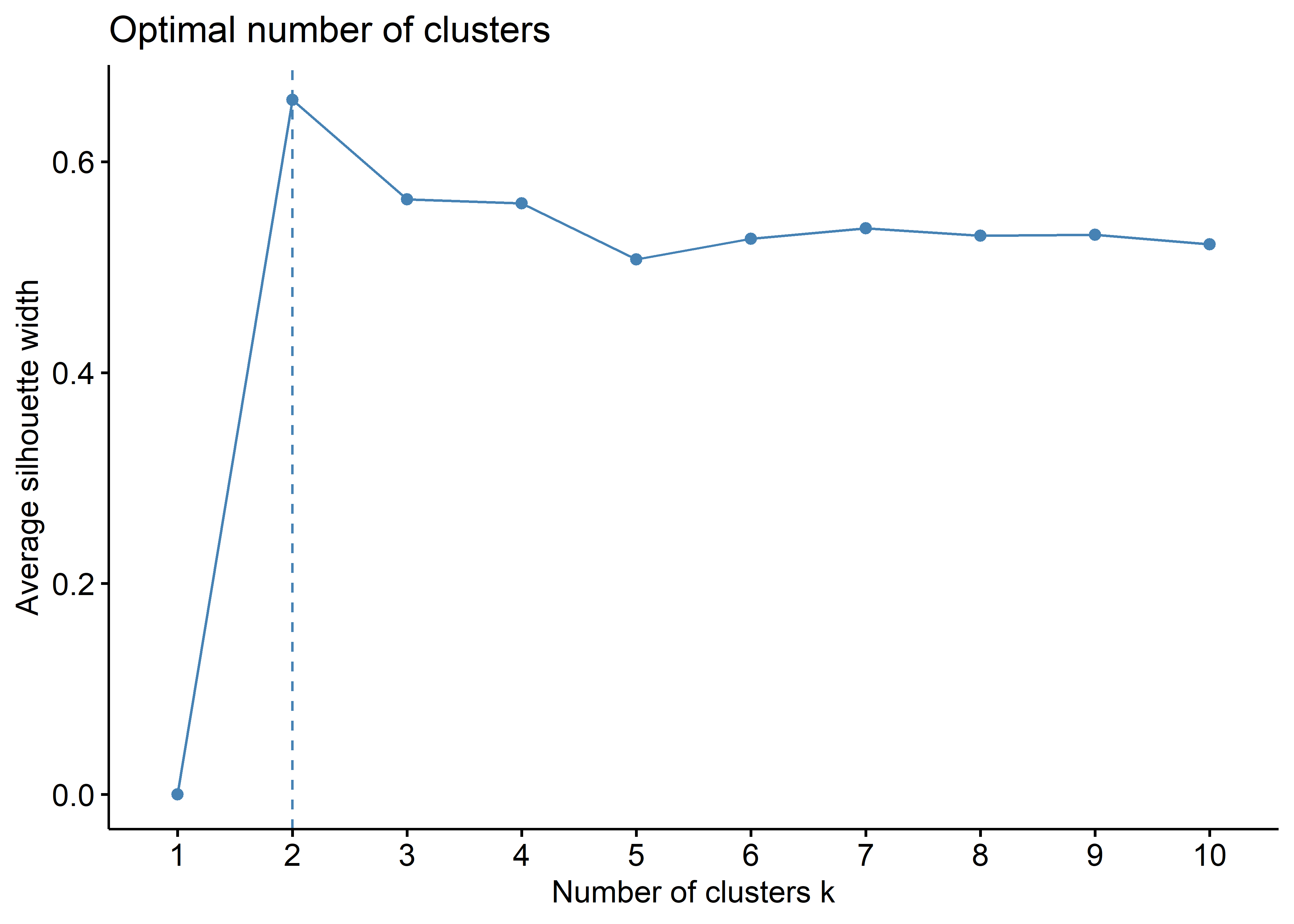
Esta gráfica nos dice que el número óptimo de clusters es 2, basado en el método average silhouette. Esta técnica, en palabras simples, mide la calidad de los clusters (i.e., qué tan bien una muestra cae dentro de un cluster).
Una vez que tenemos el número óptimo de clusters, podemos graficar nuestros resultados mediante un scatter plot. Para esto, primero cortamos el agrupamiento en dos clusters con la función cutree, y luego usamos la función fviz_cluster para hacer la gráfica:
clust_groups = cutree(my_clust, k = 2)
fviz_cluster(list(data = tmp_data, cluster = clust_groups))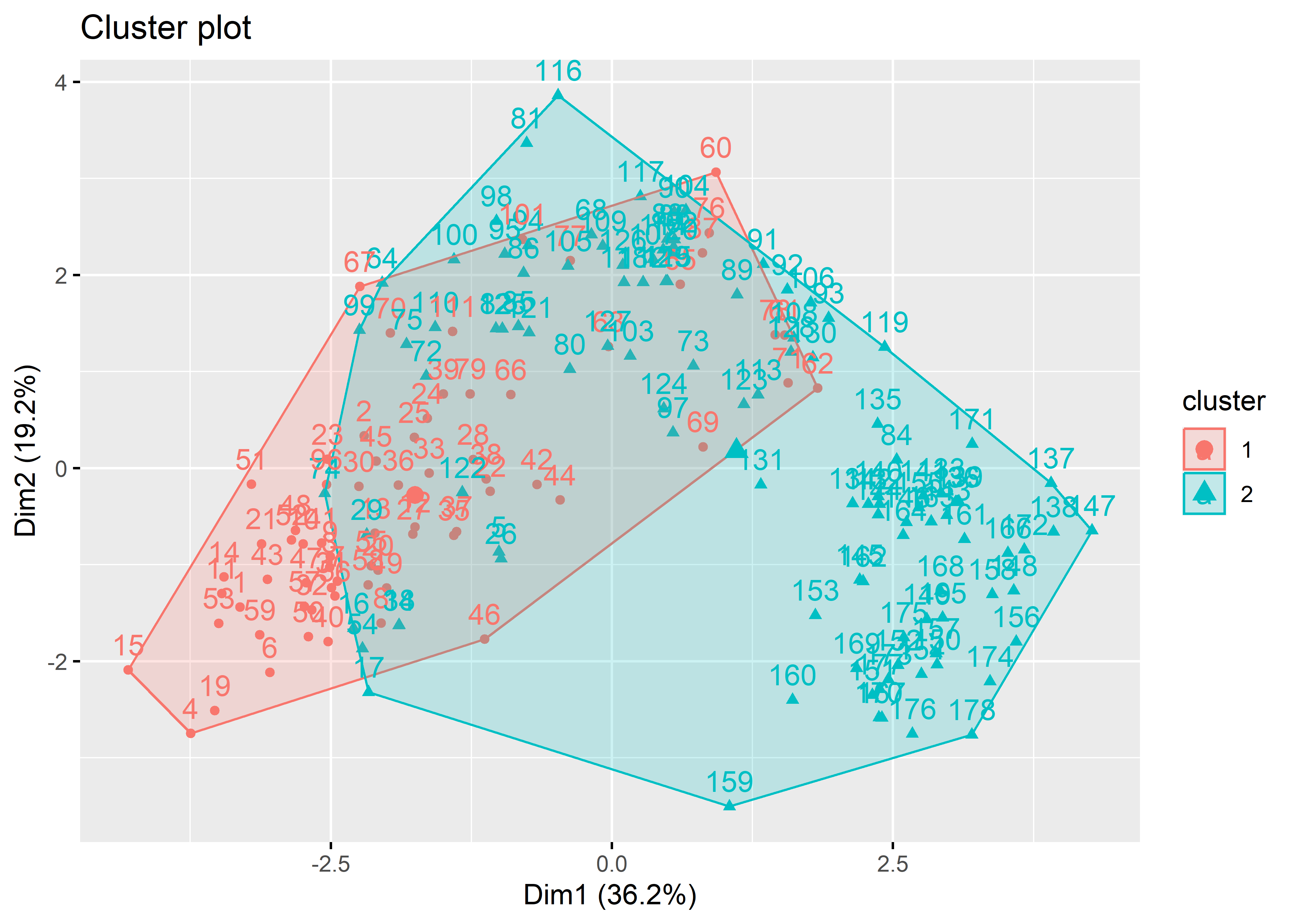
Lo que vemos en esta gráfica son las muestras agrupadas en su cluster respectivo, el cual está representado por diferentes colores.