require(tidyverse)
require(factoextra)
require(ade4)
require(GGally)Análisis de componentes principales en R
Recuerda que tenemos diversos posts sobre programación, pesquerías, ecología, entre otros. Te invitamos a revisar la lista en nuestra sección Posts.
Esta técnica nos permite visualizar y entender cómo las variables en un conjunto de datos varían, y es particularmente útil cuando trabajamos con muchas variables. Los componentes del análisis de componentes principales (ACP) nos indican las direcciones donde hay mayor varianza. Este tutorial tiene como objetivo una presentación rápida de las funciones disponibles para hacer un ACP en R.
Comenzamos cargando las librerías que utilizaremos:
Primero pasamos a explorar las bases de datos que vamos a utilizar, la cual contiene información sobre la concentración de diferentes químicos presentes en vinos de una región de Italia, derivados de 3 diferentes cultivos. Los datos y más información sobre estos puede ser encontrados aquí.
Lo primero que haremos es leer la base de datos desde el sitio web, y luego lo transformamos a clase tibble:
my_data = read.table("http://archive.ics.uci.edu/ml/machine-learning-databases/wine/wine.data", sep=",")
my_data = my_data %>% as_tibble
head(my_data)# A tibble: 6 × 14
V1 V2 V3 V4 V5 V6 V7 V8 V9 V10 V11 V12 V13
<int> <dbl> <dbl> <dbl> <dbl> <int> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 14.2 1.71 2.43 15.6 127 2.8 3.06 0.28 2.29 5.64 1.04 3.92
2 1 13.2 1.78 2.14 11.2 100 2.65 2.76 0.26 1.28 4.38 1.05 3.4
3 1 13.2 2.36 2.67 18.6 101 2.8 3.24 0.3 2.81 5.68 1.03 3.17
4 1 14.4 1.95 2.5 16.8 113 3.85 3.49 0.24 2.18 7.8 0.86 3.45
5 1 13.2 2.59 2.87 21 118 2.8 2.69 0.39 1.82 4.32 1.04 2.93
6 1 14.2 1.76 2.45 15.2 112 3.27 3.39 0.34 1.97 6.75 1.05 2.85
# ℹ 1 more variable: V14 <int>La primera columna nos indica el cultivo, y las otras 13 columnas es un químico diferente. Las filas son las muestras de vino utilizadas.
dim(my_data)[1] 178 14Como vemos, en total tenemos 178 muestras. Ahora vamos a comenzar a analizar la distribución de cada variable, y la correlación entre ellas. Para eso vamos a graficar un scatterplot, y calcular la correlación de Spearman. Solo lo haremos para los primeros 5 químicos por cuestiones didácticas, y diferenciaremos por tipo de cultivo (color):
my_data %>%
GGally::ggscatmat(columns = 2:6, color = "V1", corMethod = "spearman") +
theme(strip.background = element_rect(fill = "grey90"),
axis.text = element_text(size = 8),
legend.position = "right",
legend.key = element_blank())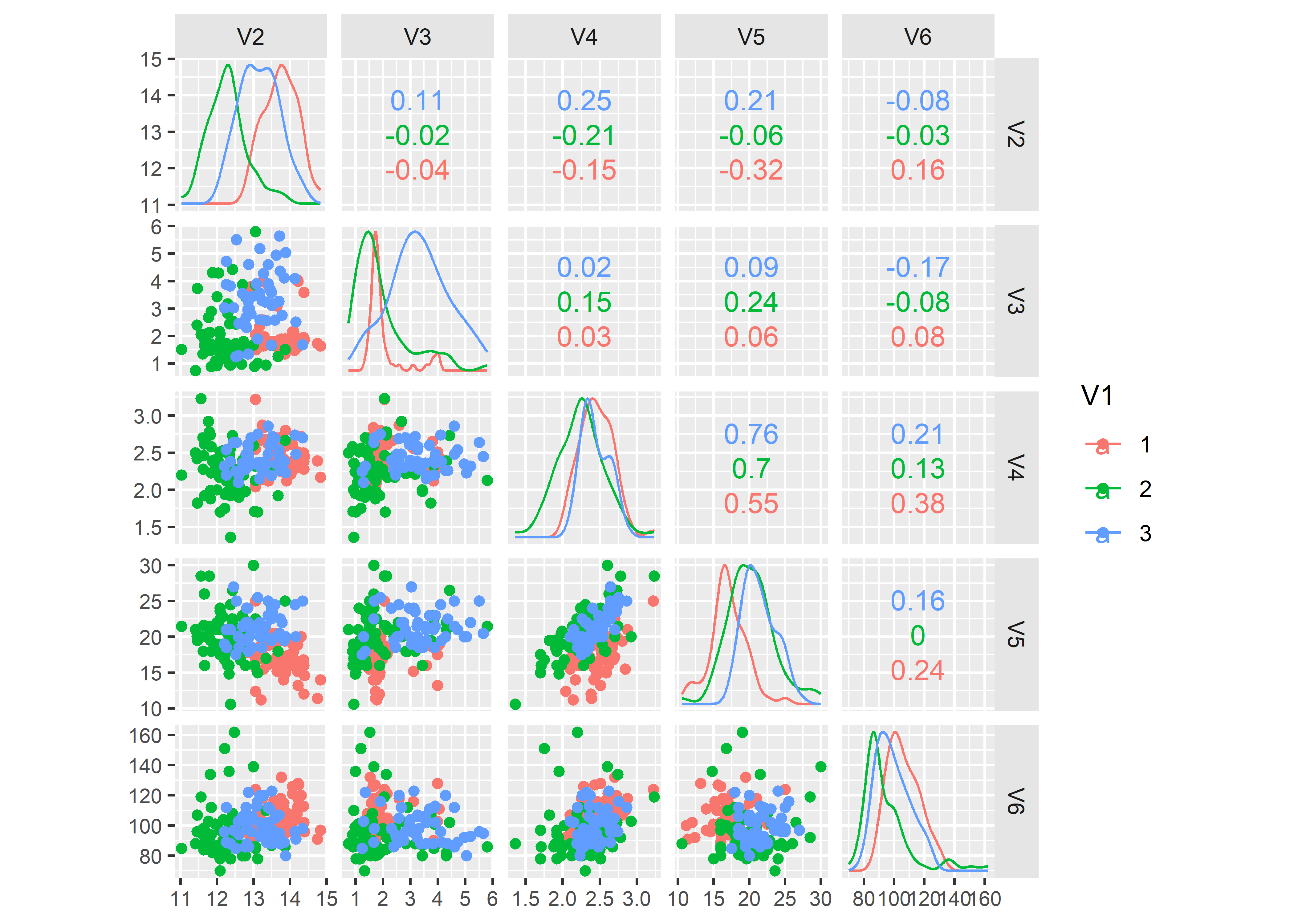
Dado que tenemos muchas variable, es díficil obtener conclusiones a partir de este análisis. Para los casos en donde trabajamos con datos multivariados como estos, es útil emplear técnicas adecuadas como un ACP. En R, podemos utilizar la función dudi.pca de la librería ade4, aunque también existen otras funciones disponibles (e.g., prcomp).
my_pca = dudi.pca(my_data, center = TRUE, scale = TRUE, scannf = FALSE)
summary(my_pca)Class: pca dudi
Call: dudi.pca(df = my_data, center = TRUE, scale = TRUE, scannf = FALSE)
Total inertia: 14
Eigenvalues:
Ax1 Ax2 Ax3 Ax4 Ax5
5.5359 2.4971 1.4461 0.9279 0.8775
Projected inertia (%):
Ax1 Ax2 Ax3 Ax4 Ax5
39.542 17.836 10.329 6.628 6.268
Cumulative projected inertia (%):
Ax1 Ax1:2 Ax1:3 Ax1:4 Ax1:5
39.54 57.38 67.71 74.34 80.60
(Only 5 dimensions (out of 14) are shown)Este resumen nos arroja información, entre otras cosas, sobre el porcentaje de la varianza en nuestros datos representados por cierto número de ejes. Por ejemplo, el eje 1 (Ax1) explica el 39.5% de la varianza en nuestros datos, el eje 2 (Ax2) explica el 17.8%, y así sucesivamente. Esto lo podemos explorar mejor con la siguiente gráfica:
fviz_screeplot(my_pca, addlabels = TRUE)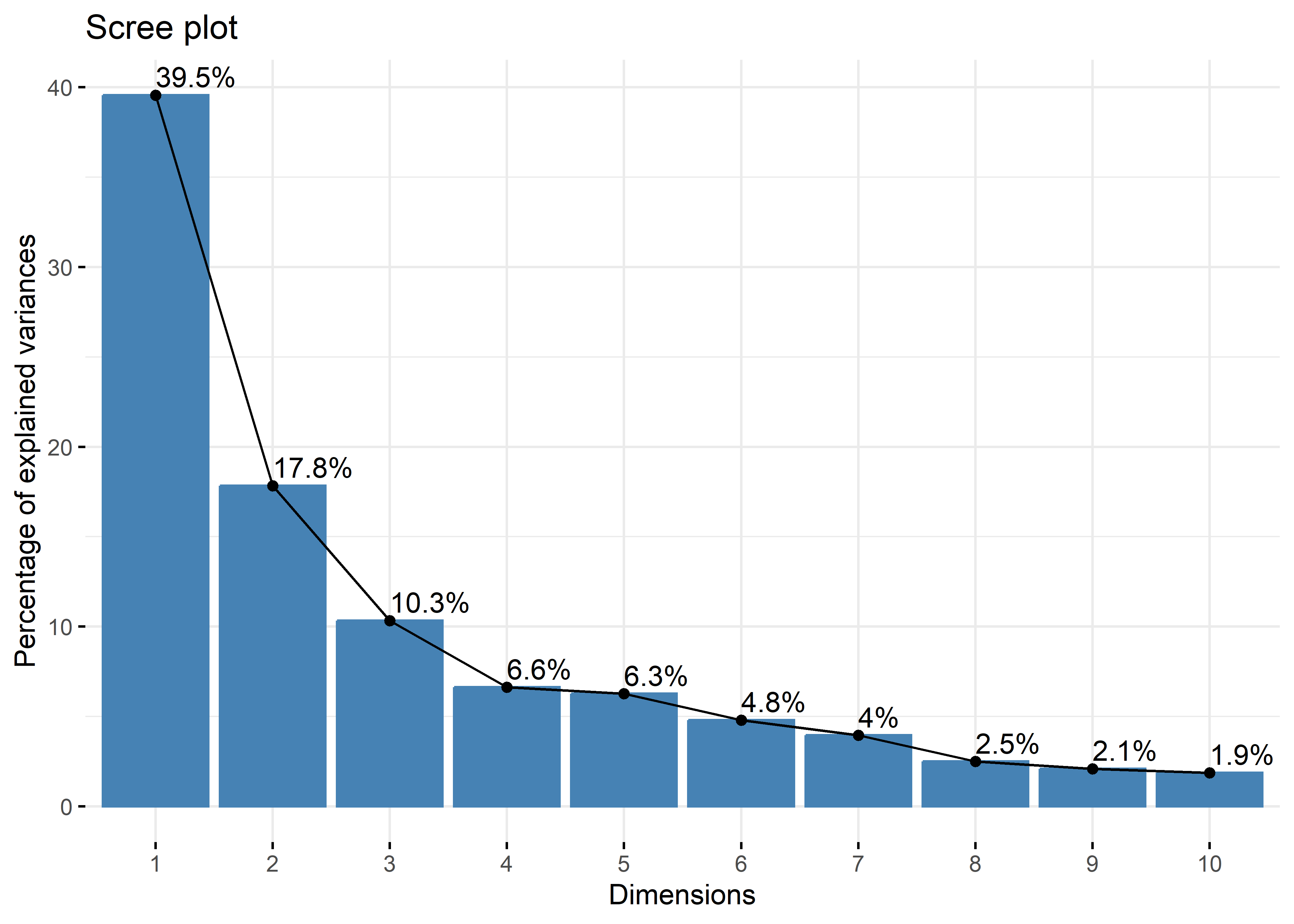
En la mayoría de casos, solemos graficar los dos primeros ejes para representar la varianza de todo el conjunto de datos. Para hacer esto, utilizamos la función fviz_pca_var de la librería factoextra:
fviz_pca_var(my_pca, col.var = "black")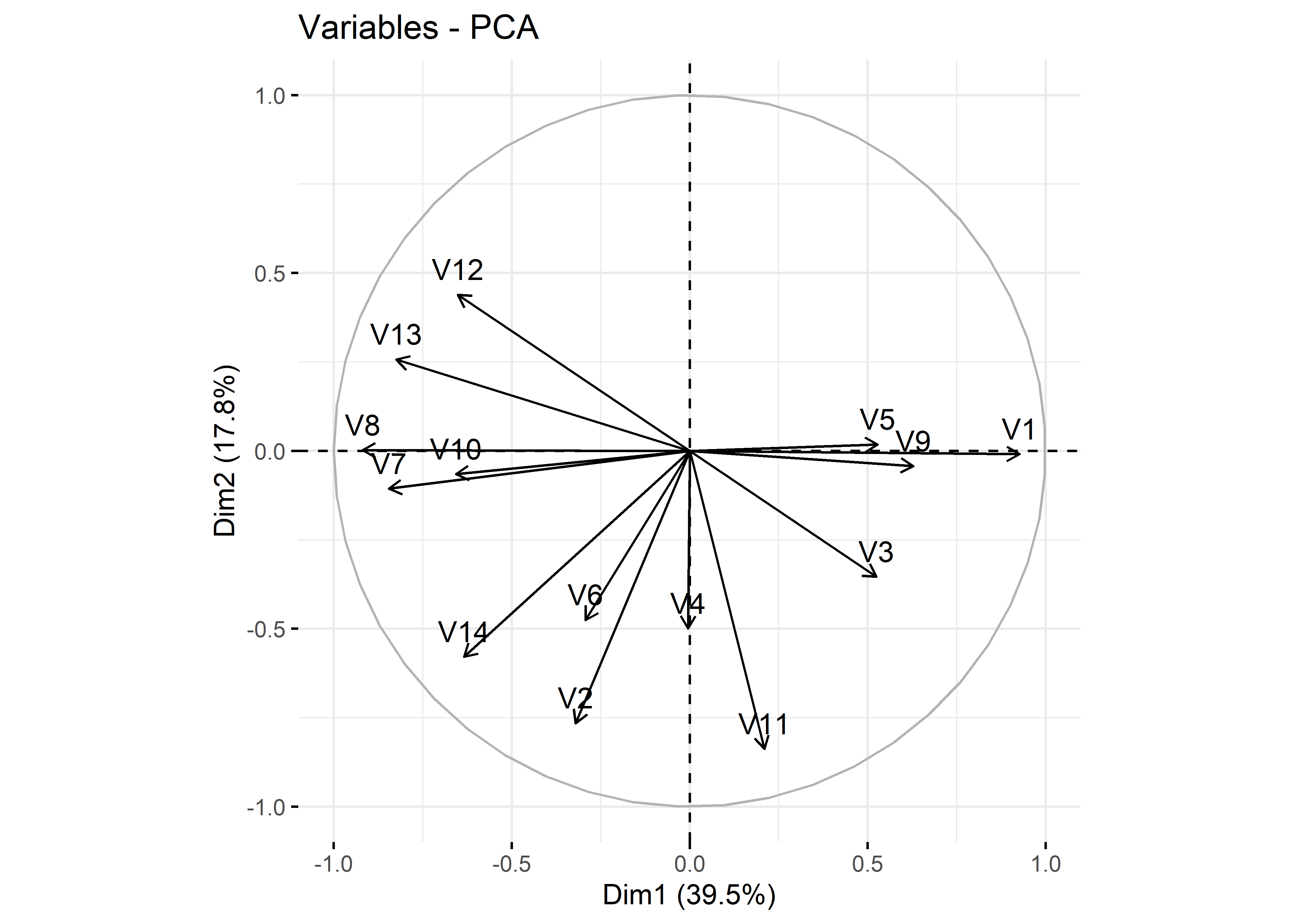
Esta gráfica nos da información sobre el grado de correlación que existe entre las variable que tenemos, en este ejemplo son los químicos en las muestras de vinos. Variables que están en la misma dirección suelen estar presentes en las mismas muestras, mientras variables que estan en direcciones opuestas no suelen estar juntas.
Finalmente, también podemos representar cada muestra en este plano, diferenciándolas por el tipo de cultivo (color):
fviz_pca_biplot(my_pca, label = "var", habillage = factor(my_data$V1),
palette = c("#00AFBB", "#E7B800", "#FC4E07"), addEllipses = TRUE)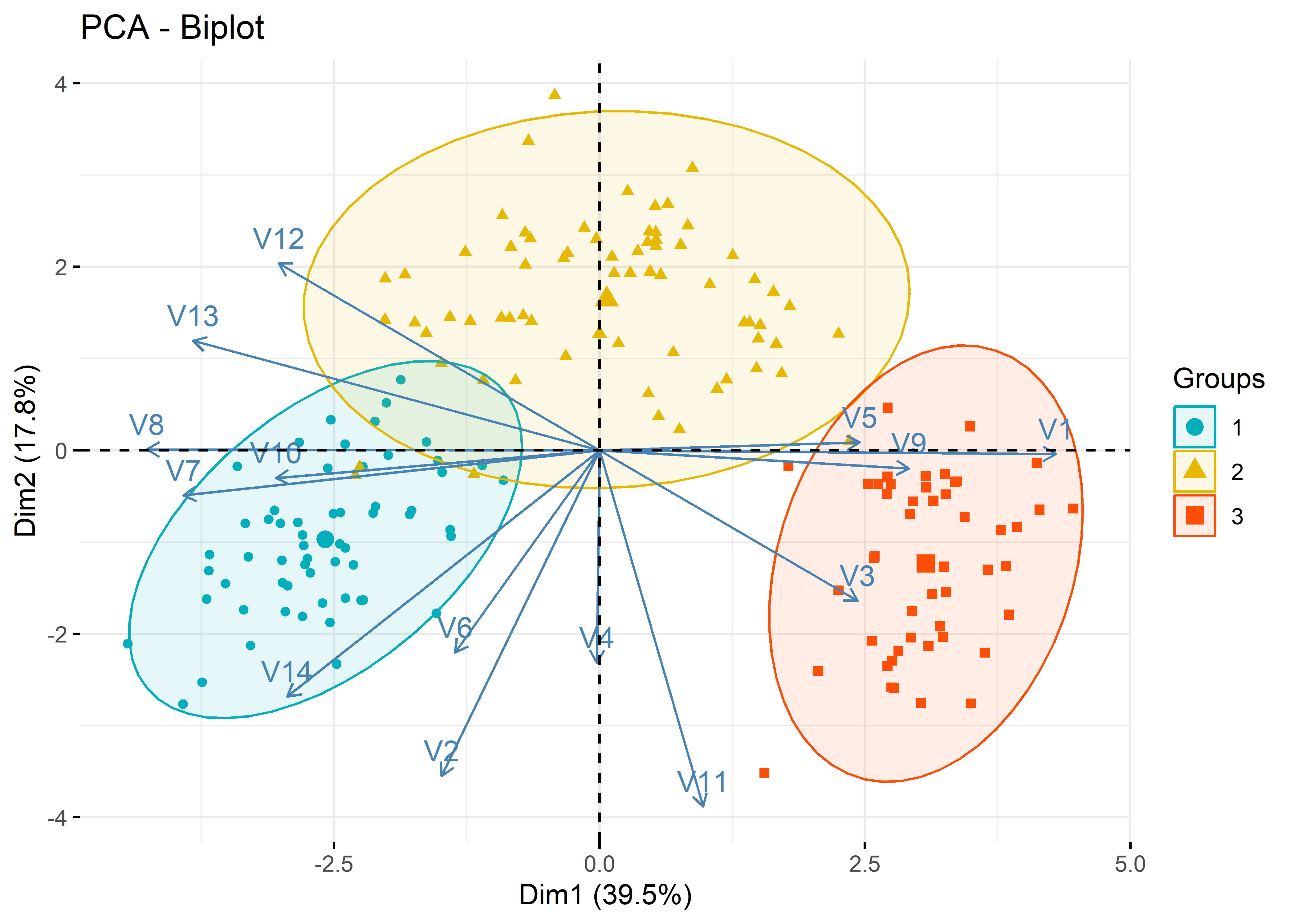
Una de las conclusiones que podríamos obtener a partir de esta gráfica es que los químicos V5, V9, Y V1 coinciden en las muestras del cultivo 3 (color naranja).